人全基因組重測(cè)序分析
產(chǎn)品簡(jiǎn)介
人全基因組測(cè)序(Whole Genome Sequencing��,WGS)是基于人基因組參考序列對(duì)個(gè)體或群體進(jìn)行全基因組測(cè)序����,并在個(gè)體或群體水平進(jìn)行分析的方法。
全基因組測(cè)序可更全面地挖掘基因序列差異和結(jié)構(gòu)變異�,包括單堿基突變、插入缺失變異����、拷貝數(shù)變異和結(jié)構(gòu)變異���,可用于基因多樣性分析、遺傳進(jìn)化分析以及致病和易感基因篩選等���,已成為人類(lèi)遺傳學(xué)��、轉(zhuǎn)化醫(yī)學(xué)和群體進(jìn)化領(lǐng)域最為迅速而有效的方法之一���。
技術(shù)路線(xiàn)
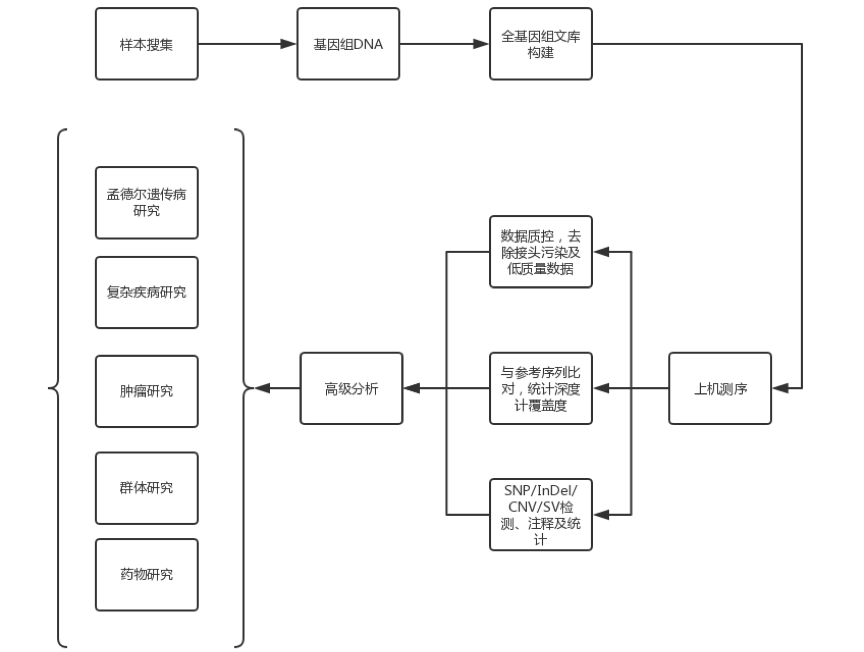
分析內(nèi)容
|
標(biāo)準(zhǔn)分析
|
1. 數(shù)據(jù)質(zhì)控:去除接頭污染和低質(zhì)量數(shù)據(jù)
2.與參考序列進(jìn)行比對(duì)、統(tǒng)計(jì)測(cè)序深度及覆蓋度
3. SNP/InDel/CNV /SV檢測(cè)�����、注釋及統(tǒng)計(jì)
4.基因組變異 Circos 圖
|
|
高級(jí)分析
|
(一)基于變異有害性的篩選
1. 突變位點(diǎn)篩選
(1)篩選的突變過(guò)濾已知數(shù)據(jù)庫(kù)�����;
(2)篩選的變異保留編碼區(qū)或剪切位點(diǎn)區(qū)的變異位點(diǎn)�����;
(3)氨基酸保守性預(yù)測(cè)
2.突變位點(diǎn)有害性分類(lèi)(ACMG)
3.Non-coding 區(qū)突變位點(diǎn)篩選
4.結(jié)構(gòu)變異 CNV/SV 有害性分析
(二)基于選樣信息的篩選
1.顯性/隱性遺傳模式分析(需提供家系圖)
1.1 顯性遺傳模式
1.2 隱性遺傳模式
2.家系連鎖分析(家系樣本)
3.純合子區(qū)域(ROH)分析(近親結(jié)婚家系樣本)
4.共有突變基因篩選(散發(fā)樣本)
(三)基于基因功能和表型的篩選
1.候選基因功能富集分析
2.候選基因通路富集分析
3.候選基因與疾病相關(guān)性排序
(四)個(gè)性化分析
1. GWAS分析
2. 其他個(gè)性化分析
|
案例展示
Whole genome sequencing resource identifies 18 new candidate genes for autism spectrum disorder
全基因組測(cè)序鑒定出18個(gè)新的自閉癥譜系障礙候選基因
加拿大多倫多的研究團(tuán)隊(duì)在Nature·Neuroscience上發(fā)表文章�����,公布了最大的自閉癥譜系障礙全基因組測(cè)序數(shù)據(jù)����。
研究人員搜集了5205個(gè)自閉癥樣本,通過(guò)不同測(cè)序平臺(tái)進(jìn)行全基因組測(cè)序�����。測(cè)序發(fā)現(xiàn)每個(gè)個(gè)體平均鑒定出73.8 個(gè)新的單核苷酸突變����,12.6 個(gè)新的InDels 或拷貝數(shù)變異。經(jīng)過(guò)與數(shù)據(jù)庫(kù)比對(duì)分析�,鑒定出61 個(gè)泛自閉癥障礙的風(fēng)險(xiǎn)基因,其中包含18 個(gè)沒(méi)有被報(bào)道過(guò)的基因����。攜帶這些基因變異的患者有較低的社會(huì)適應(yīng)能力。本研究通過(guò)大樣本的全基因組測(cè)序�,為自閉癥的后續(xù)研究、分子診斷及藥物開(kāi)發(fā)提供了基礎(chǔ)�����。
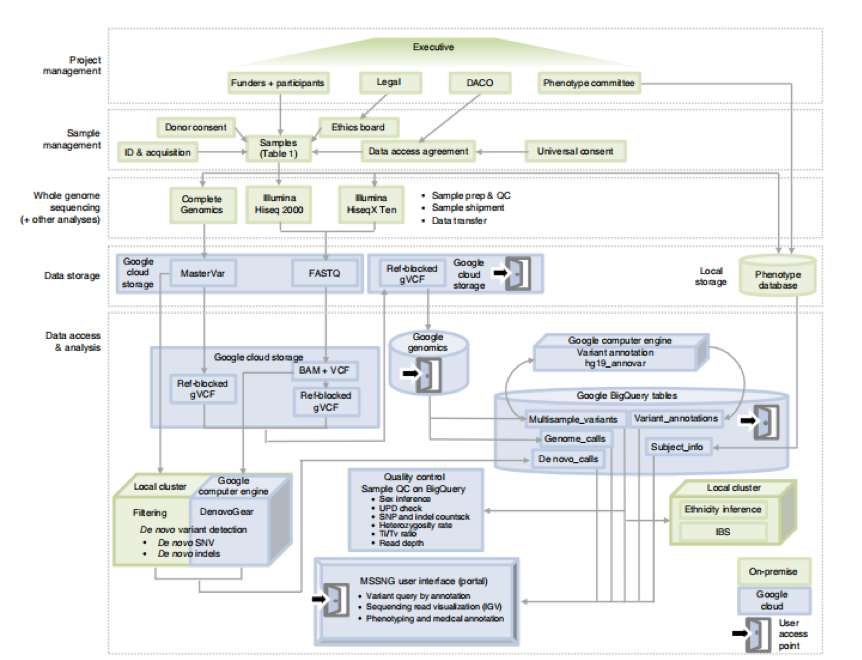
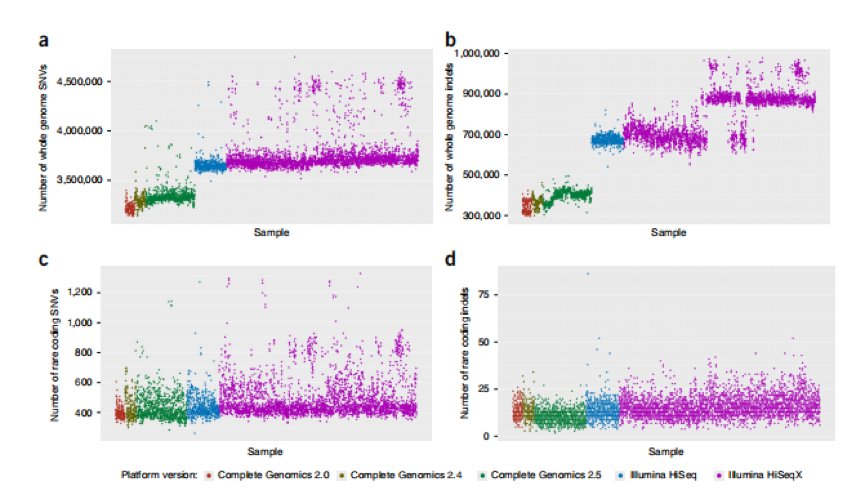
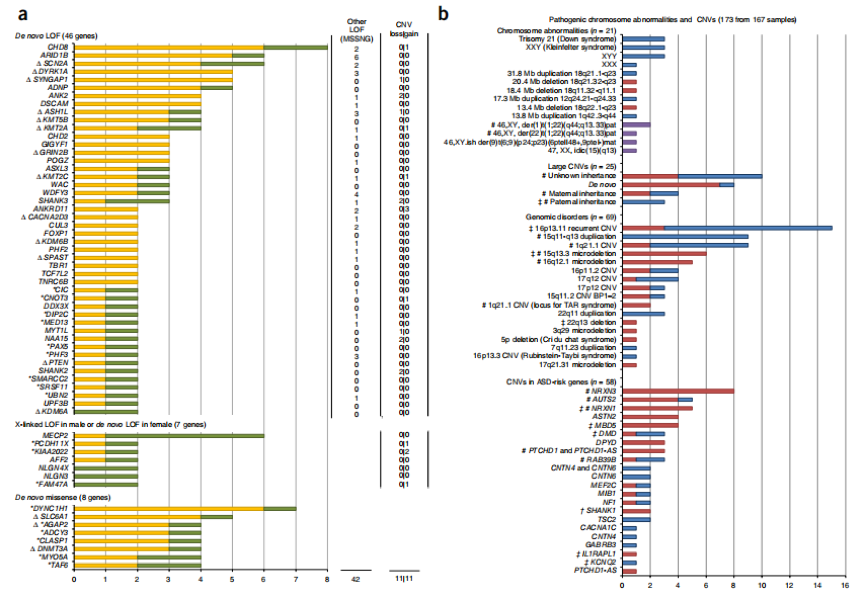
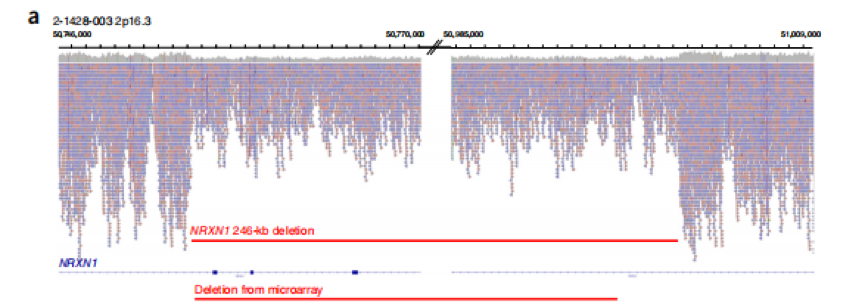
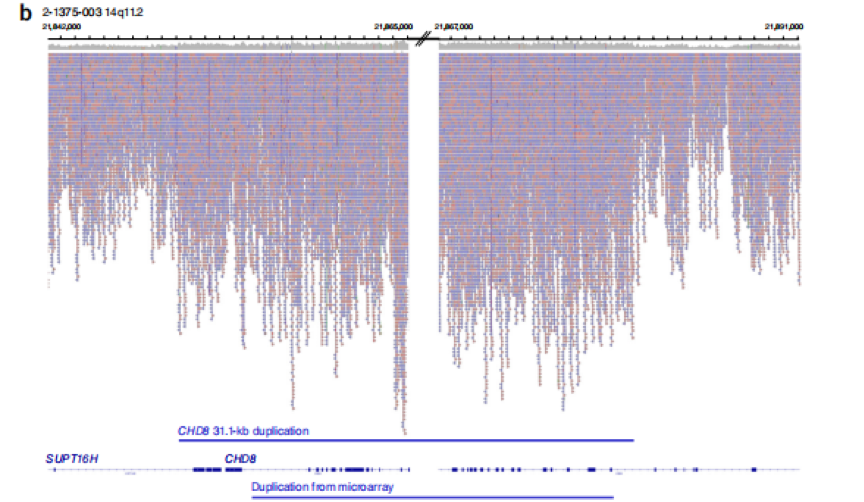


C Yuen Ryan K,Merico Daniele,Bookman Matt et al. Whole genome sequencing resource identifies 18 new candidate genes for autism spectrum disorder.[J] .Nat. Neurosci., 2017, 20: 602-611.


 韋翰斯生物公眾號(hào)
韋翰斯生物公眾號(hào)

 滬公網(wǎng)安備31011502401676
滬公網(wǎng)安備31011502401676